近日,上海交通大学自然科学研究院与物理与天文学院的徐恒课题组与其合作者,在Nature Communications上发表了题为“Uncovering dynamic transcriptional regulation of methanogenesis via single-cell imaging of archaeal gene expression”的研究论文。该研究首次在单细胞尺度上揭示了古菌产甲烷代谢通路的动态转录调控动力学机制,为深入理解古菌甲烷代谢过程提供了新的视角。
研究背景
生物体的新陈代谢为了适应不断变化的环境和生长需求,其基因转录时刻受到精确调控。这种调控不仅优化了群体水平的代谢进程,同时也能导致细胞间的显著异质性,从而提高群体在复杂环境中的资源利用效率与适应能力。因此,在单细胞尺度上定量解析代谢通路的转录调控动力学机制,对理解代谢过程具有重要的科学价值。
目前,单细胞水平的代谢调控研究主要集中在真核生物和细菌,而对生命的第三个域——古菌的研究仍十分有限。由于古菌独特的细胞结构及其较低的 mRNA 含量,现有技术难以实现对单个古菌细胞基因表达的精确测量,这极大地限制了我们对各种具有重要意义的古菌代谢过程的理解。例如,古菌的产甲烷代谢在全球碳循环和气候变化中发挥着重要的作用,其代谢过程涉及一系列复杂的酶促反应,并受到多种细胞信号与环境因子的调控。然而,由于现有研究大多基于宏观测量方法,难以揭示产甲烷代谢通路在单细胞尺度上的动态运行机制,及其对关键环境因子的响应模式。
研究方法及结果
在本工作中,研究人员基于多轮杂交链式反应—单分子荧光原位杂交技术(smHCR-FISH),开发了一种适用于古菌的单细胞、单分子定量成像方法(图1A)。利用该方法,研究人员以一种典型的甲基营养型产甲烷古菌——东方产甲烷球菌(Methanococcoides orientis, LMO-1)为例,定量测量了产甲烷代谢通路中的12种关键功能基因的单细胞转录动力学(图1B)。研究发现,这些关键代谢基因的表达呈现出特定的时序模式,与产甲烷代谢过程中酶促反应的顺序相匹配(图1C)。此外,研究还发现,一种重要的环境因子Fe(III)能够改变上述基因的表达模式,并通过非酶促的芬顿反应显著提高甲烷产率(图1C)。通过对大量细胞进行单细胞聚类分析,研究人员发现,生长在相同的环境条件下的产甲烷古菌种群中存在多个具有独特基因表达和时间动态模式的代谢亚群(图1D),而Fe(III)的存在则会导致这些代谢亚群的时序发生重排。进一步的单细胞动力学分析和生物物理学建模表明,产甲烷代谢相关基因的转录过程遵循爆发式动力学模式,且不同基因的转录调控可能依赖几种共同的调控因子(图1E)。
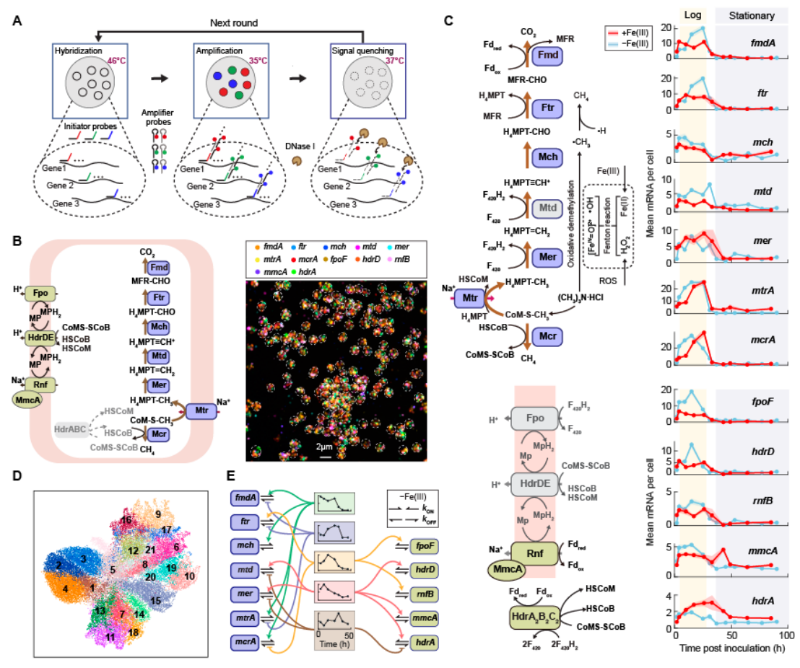
图1. (A)多轮smHCR-FISH方法示意图。(B) 产甲烷古菌LMO-1的产甲烷代谢通路以及甲烷代谢关键基因的单细胞成像。(C) 产甲烷代谢关键基因的表达随细胞生长时间的变化曲线。(D)通过UMAP可视化展示LMO-1群体中的多个代谢亚群。(E)产甲烷代谢关键基因的转录调控模式。
本研究工作为理解古菌产甲烷代谢的转录调控机制提供了关键信息,并为深入研究其他古菌或复杂微生物菌落的代谢调控机制提供了新的技术手段和研究思路。
上海交通大学物理与天文学院博士生董怡静为论文第一作者,上海交通大学徐恒副教授和上海交通大学海洋学院王风平教授为共同通讯作者。上海交通大学硕士生漆兰婷、博士生赵菲、陈一凡以及梁乐文博士、王景博士和赵维殳副研究员为文章的合作作者。本研究得到了国家自然科学基金、科技部重点研发计划以及上海市自然科学基金的资助。
作者: 自然科学研究院 供稿单位: 自然科学研究院
© 版权声明
本文由分享者转载或发布,内容仅供学习和交流,版权归原文作者所有。如有侵权,请留言联系更正或删除。
相关文章

暂无评论...